IDENTIFICACIÓN DE HONGOS, OOMYCETOS, PLANTAS E INSECTOS MEDIANTE RECONSTRUCCIÓN FILOGENÉTICA
36o. Curso en línea
4, 5 y 6 de febrero de 2026
Impartido por:
Dra. Hilda Victoria Silva Rojas
Colegio de Postgraduados

Dirigido a:
A estudiantes, personal de laboratorios e investigadores relacionados con la identificación de microorganismos, plantas e insectos.
Objetivo:
Introducir a los participantes en los principales métodos y herramientas bioinformáticas, utilizadas para la construcción de árboles filogenéticos que permitan la identificación correcta de organismos vivos y el reconocimiento de nuevas especies.
Programa del Curso:
MIÉRCOLES 4 DE FEBRERO | 4:00 – 7:00 pm
Bienvenida al curso
- Identificación de organismos a través de reconstrucción filogenética.
- Ensamble de secuencias de DNA (Bioedit).
JUEVES 5 DE FEBRERO | 4:00 – 7:00 pm
- Alineamiento múltiple de secuencias (MAFFT y Mega12).
- Modelos de sustitución nucleotídica: Selección de modelos (jModelTest).
- Concatenado de secuencias de genes MLST (Mesquite).
VIERNES 6 DE FEBRERO | 4:00 – 7:00 pm
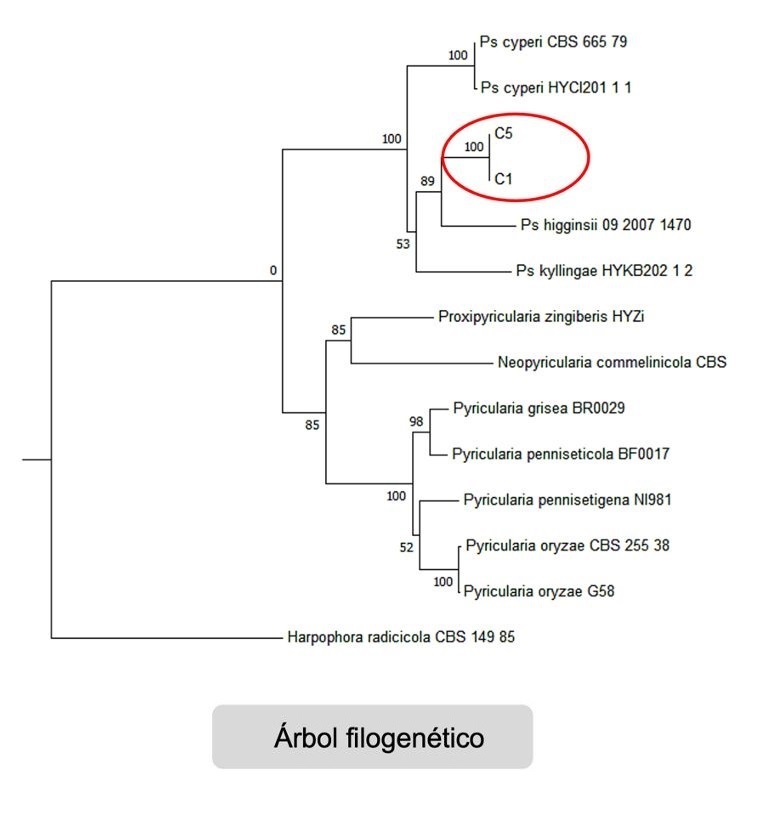
- Reconstrucción filogenética: Construcción de árboles filogenéticos mediante inferencia bayesiana (Mr Bayes) y máxima verosimilitud (RaxML).
- Visualización de árboles filogenéticos (FigTree).
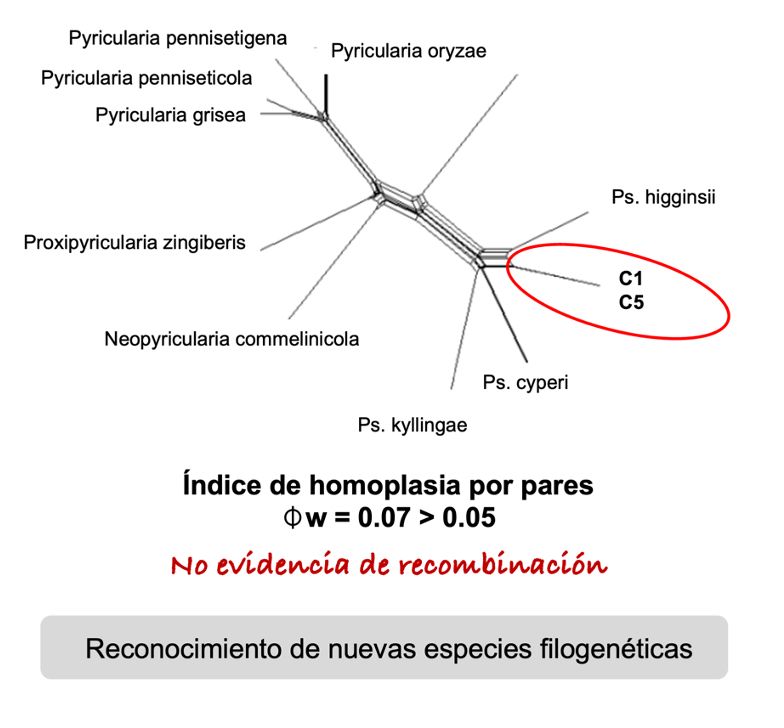
- Reconocimiento de especies filogenéticas por concordancia genealógica, cálculo del índice de homplasia por pares (Splitstree4).
Horario:
De 4:00PM a 7:00PM (CDMX)
Se extenderá constancia de participación con una duración de 10 horas.
Plataforma:
BVDash & Zoom (Recibirás la información y enlace de acceso al curso dos (2) días antes de la fecha de inicio del mismo).
Inscripción:
Costo: US$180
Para más información por favor escribanos al siguiente correo electrónico:
support@bvdash.com